Ich hatte es ja schon länger versprochen, dass ich mich daran mache, das eigentliche Thema meiner Arbeit genauer zu Erläutern. Der Arbeitstitel lautet erstmal:
‚Experimental and theoretical analysis of estrogen receptor transcriptional cycling‘
Zur Gentranskription habe ich ja kürzlich schon einen allgemeinen Artikel geschrieben. In meiner Arbeit soll es nun um einen Teilaspekt dessen gehen. Nämlich die Regulation des ps2 Genes, das über Östrogenrezeptoren reguliert wird. Um diese Regulation zu erfassen, schaut man sich an, zu welchem Zeitpunkt welche Transkriptionsfaktoren an den ps2 Promoter gebunden sind.
Östrogene sind die weiblichen Gechlechtshormone und bestimmte Gewebe sind sensitiv für Östrogen, d.h. sie ändern ihr Verhalten, wenn damit stimuliert. Die Brustdrüsen sind ein solches Gewebe. MCF7 Zellen sind Brustkrebszellen und Estradiol-sensitiv, d.h. sie reagieren auf Estradiol (E2, eine Östrogenform). Dazu haben sie Östrogenrezeptoren, an die E2 binden kann und in gebundener Form fungieren die Rezeptoren als Transkriptionsfaktoren, genauer als Aktivatoren der Transkription des ps2 Gens. Rezeptoren für E2 sind ER und ER
aus der Klasse der Nuklear- oder Kernrezeptoren. Kernrezeptoren deshalb, weil sie im Zellkern vorkommen. Diese Rezeptoren agieren als Transkriptionsfaktoren. ER
ist interessant für meinen Fall, da er, wenn E2 gebunden, die Transkription des ps2 Gens aktiviert. Allerdings sind dafür noch eine ganze Menge andere Proteine nötig, wie ich vorher schon beschrieben habe.
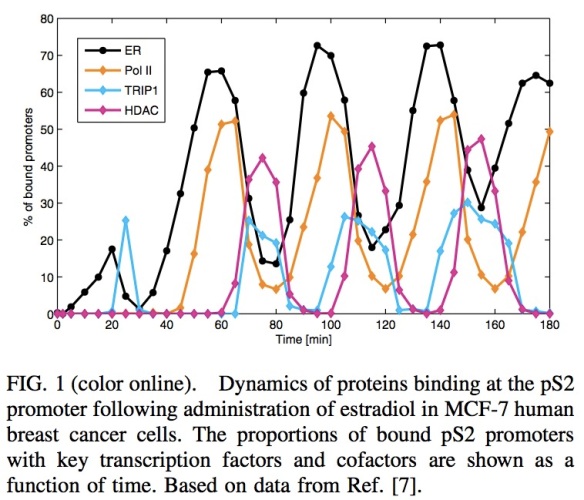
Die Transkription selber erfolgt dann in einem zyklischen Muster (siehe Bild) mit einer Periodendauer von etwa 40 Minuten. Dargestellt sind der Prozentsatz der Promoter, die vom jeweiligen Protein gebunden sind über die Zeit. Die Messungen wurden durch Chromatin Immunopräzipitationsexperimente gewonnen, denen ich einen eigenen Artikel widmen werde um euch auch an meinen Labor(miss)erfolgen teilhaben zu lassen.
Die schwarze Kurve stellt den Verlauf der Östrogenrezeptoren dar, die an den Promoter binden und orange repräsentiert den der RNA Polymerase II. Nur wenn letztere am Promoter gebunden ist, kann Transkription überhaupt stattfinden. Das führt dann zu Transkriptionszyklen und damit auch zum Namen meines Blogs.
Das spannende an der Geschichte ist nun, der hohe Grad von Ordnung in diesem Prozess. Métivier et al. haben für ihren Artikel von 2003 diese Zeitserien nicht nur für 4 Transkriptionsfaktoren gemessen, sondern für über 40. Das muss nicht nur wahnsinnig viel experimentelle Arbeit gewesen sein, sondern weist auch darauf hin, dass es eine ziemlich genau festgelegte Abfolge der Ereignisse geben muss. Durch weitere Re-ChIP experimente, wo sie auf Kombinationen der Transkriptionsfaktoren testeten, konnten sie eine Reihenfolge der Bindungsereignisse postulieren. Grob könnte man es so beschreiben, dass erst die DNA aufgewickelt werden muss, sprich zugänglich gemacht werden, dann kommt die basale Transkriptionsmachinerie und dazu die RNA pol II. Am Ende des Zyklus werden die ganzen Transkriptionsfaktoren wieder vom Promoter abgeräumt und ein neuer Zyklus kann beginnen. In ihrem späteren EMBO report bezeichen sie das als eine Art ‚Transkriptionsratsche‘, also einen Prozess, der nur in eine Richtung abläuft, also eine wohldefinierte Richtung hat. Allerdings hat der Zyklus nicht für alle Teilnehmer die gleiche Periode. Manche Transkriptionsfaktoren bleiben auch über zwei Zyklen der RNA pol II gebunden, sie müssen also bis mindestens zwei zählen können.
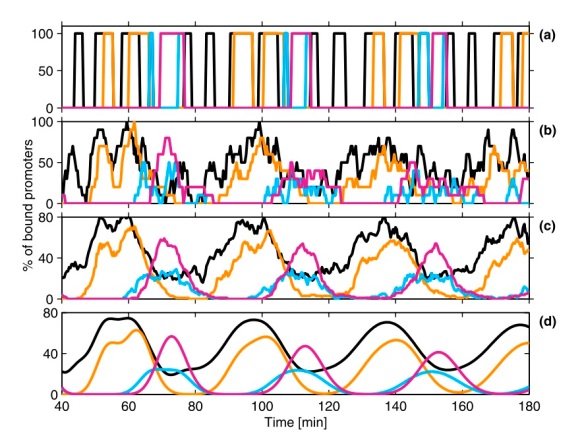
Die ChIP Experimente sind immer eine Ensemblemessung, d.h. sie messen die Besetzung des Promoters in sehr vielen Zellen. Man braucht also sehr viele Zellkulturen (für jeden zu messenden Zeitpunkt eine) und erhält durch jede immer eine Momentaufnahme. Allerdings ist Gentranskription ein Vorgang, der in jeder Zelle abläuft, wenn man diese Zyklen verstehen will, muss man sich überlegen, was auf Einzelzellebene abläuft und wie daraus in synchronisierten Zellen die obigen Muster entstehen können.
Ein schönes konzeptionellen Paper haben dazu Lemaire et al. publiziert. Sie haben sich Gedanken gemacht, wie aus stochastischen Vorgängen in einer einzelnen Zelle diese Zyklen entstehen können. Im Prinzip ist ja jedes Binden eines Transkriptionsfaktors ein binäres Ereignis, das auch noch stochastisch erfolgt.
Sie haben also unter bestimmten Annahmen (Anzahl der Reaktionsschritte, einzelne Reaktionsraten) simuliert, was auf Einzelebene passiert und dann geschaut, wie es aussieht, wenn man es für 10 oder 100 Zellen simuliert. Man erhält, wenn auch noch recht verrauscht, ein periodisches Muster. In einer Zellkultur sind nun allerdings etwa eine Million Zellen, was dann zu einer glatteren Kurve führen sollte. Das spannende an diesem Paper ist, dass es Einzelzellereignisse mit dem Ensembleverhalten verbindet und so eine interessante Methode aufzeigt das Verhalten zu modellieren. Denn das ist ja das Ziel meiner Arbeit, ein mechanistisches Modell der Transkriptionszyklen zu finden und am Experiment zu testen.
Einer der Autoren der zitierten Artikel, George Reid, arbeitet hier am IMB und unter anderem auch an diesen Transkriptionszyklen. Mit ihm zusammen plane ich meine Experimente und Iryna (sie ist Post Doc in Georges Gruppe) ist meine Lehrerin in Sachen Molekularbiologie und im Moment nehme ich sie mit meinen vielen Fragen ganz schön in Beschlag.
Ich möchte also für die Zukunft die oben kurz vogestellte Modellierungsmethode verfeinern und neuere experimentelle Erkenntnisse mit einbauen (wie z.B. den unproduktiven ersten Zyklus und den doppelt so langen Zyklus einzelner Transkriptionsfaktoren) und natürlich auch eigene Experimente machen. Vor allem letzteres wird sicher einige Zeit brauchen, zum einen sind diese ChIP Experimente ohnehin sehr zeitaufwändig und für den Anfänger wie mich noch mehr und zum anderen geht bei solchen Experimenten an biologischen Systemen gerne mal etwas schief. Ich werde euch auf dem laufenden halten.
Referenzen:
R. Métivier, G. Penot, M.R. Hübner, G. Reid, H. Brand, M. Kos, F. Gannon, Estrogen receptor-alpha directs ordered, cyclical, and combinatorial recruitment of cofactors on a natural target promoter, Cell (2003)
V. Lemaire, C. F. Lee, J. Lei. T. Métivier and L. Glass, Sequential recruitment and combinatoriel assembling of multiprotein complexes in transcriptional activation, Phys. Rev.Let. (2006)
R. Métivier, G. Reid, F. Gannon, Transcription in fur dimensions: nuclear receptor-directed initiation of gene expression, EMBO reports (2006)